Machen unsere Gene uns schlau? Über die molekularen Grundlagen der Intelligenz
Schul- und Berufserfolg, eine gesunde Lebensweise und Lebenserwartung hängen von der allgemeinen Intelligenz ab. Die Entschlüsselung jeglicher Ursachen von Intelligenzunterschieden ist daher von einem hohen wissenschaftlichen, aber auch gesellschaftlichen Interesse. Adoptions- und Zwillingsstudien haben wiederholt gezeigt, dass Anlage und Umwelt in etwa gleichen Teilen Intelligenzunterschiede bedingen. Dass Lebenserfahrung und Schulbildung, wie beispielsweise Problemlösefähigkeiten, die Grundlagen der Mathematik oder Prinzipien der formalen Logik, zur Entwicklung und Entfaltung der Intelligenz beitragen, lässt sich intuitiv nachvollziehen. Doch wie kann es sein, dass unser genetisches Erbe einen Einfluss auf unsere kognitive Leistungsfähigkeit hat?
Intelligenz und intelligentes Handeln entspringt dem menschlichen Gehirn. Um zu verstehen, wie unser genetisches Erbe die Intelligenz beeinflusst, muss man sich bewusst machen, was genau genetische Unterschiede sind und wie sie auf das menschliche Gehirn wirken. Dieser eher allgemeinen Frage schließen sich wesentlich konkretere an: Wenn genetische Faktoren so einen starken Einfluss auf allgemeine kognitive Funktionen haben, müsste es auch möglich sein, einzelne Gene zu identifizieren, die ihren Beitrag zur Intelligenzunterschieden leisten. Wie heißen die Gene, wo liegen sie auf unserer DNA und welche Rolle spielen sie im menschlichen Gehirn?
Das Genom: Der Bauplan von Körper und Geist
Das menschliche Genom umfasst 23 Chromosomenpaare, die aus der DNA mit der gesamten genetischen Information bestehen und im Zellkern jeder Körperzelle vollständig vorliegen. Die DNA selbst ist eine lange Abfolge von Nukleotiden, die von einem Zucker und einer Phosphatgruppe zusammengehalten werden und eine von vier verschiedenen Basen enthalten. Diese Basen heißen Adenin, Guanin, Cytosin und Thymin und bilden die Buchstaben unseres genetischen Codes. Abbildung 1 zeigt einen kleinen Ausschnitt der (doppelsträngigen) DNA. Die Buchstaben A, G, C, und T kennzeichnen die verschiedenen Basen. Auf der DNA liegen funktionale Abschnitte vor, die Gene genannt werden. Ein Gen ist eine definierte Abfolge von Basen, die den Bauplan für ein Eiweißmolekül stellen. Eiweißmoleküle geben den Zellen unseres Körpers ihre Struktur, übernehmen allerdings auch wichtige Aufgaben beim Ablauf biochemischer Reaktionen und leisten somit einen wesentlichen Beitrag zum Stoffwechsel und damit auch zur Funktion der Körperzellen. Bekannte Beispiele für solche Eiweißmoleküle sind Kollagene, die das Bindegewebe des menschlichen Körpers bilden, und das Insulin, das als Hormon den Blutzuckerspiegel reguliert. Insgesamt liegen auf der menschlichen DNA in etwa 23 000 Gene.
Kein Mensch ist wie der andere: Genetische Vielfalt
Eine der zentralen Erkenntnisse des Human Genome Projects, das mit der vollständigen Entschlüsselung des menschlichen Erbguts im Jahre 2003 erfolgreich abgeschlossen wurde, ist, dass sich die Erbinformation zweier zufällig ausgewählter Menschen unabhängig von ihrer Herkunft kaum unterscheidet. Das heißt, dass die Abfolge von Basen auf der
DNA und damit die Baupläne für Eiweißmoleküle nahezu identisch sind. In Zahlen ausgedrückt sind 99.9 % unserer Nukleobasen identisch. Dies bedeutet im Umkehrschluss, dass zwei unverwandte Menschen sich lediglich in 0.1 % aller Basen unterscheiden und dass es mit Hinblick auf die Suche nach genetisch bedingten Intelligenzunterschieden diese 0.1 % sind, in denen sich relevante Information verbirgt. Doch auch wenn sich dies zunächst überschaubar anhören mag: Aktuell listet die biomedizinische Datenbank dbsnp 60 742 281 bekannte Einzelnukleotidpolymorphismen (Stand: Juni 2014). Einzelnukleotidpolymorphismen (SNPs, sprich „Snipp“, von der englischen Bezeichnung „single nucleotide polymorphism“) bezeichnen Stellen des Genoms, die in verschiedenen Formen (polymorph) vorliegen. SNPs stellen die einfachste und häufigste Form genetischer Unterschiede dar, zeichnen sich für den Großteil der oben erwähnten 0.1 % verantwortlich und sind daher oftmals das Ziel molekulargenetischer Forschung in der Psychologie. Verschiedene Formen eines SNPs unterscheiden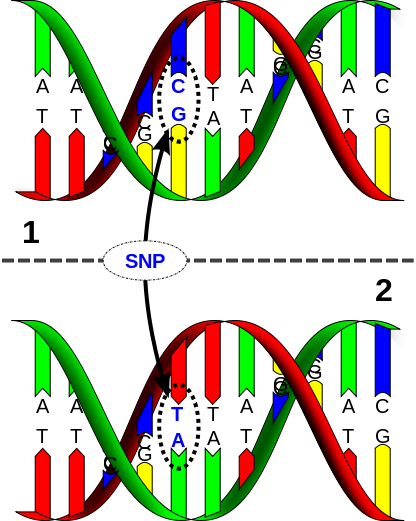 Abbildung 1: Darstellung eines Einzelnukleotipolymorphismus (SNP). Zu sehen ist die doppelsträngige DNA in der bekannten Doppelhelixform. Der DNA Abschnitt in der oberen und unteren Abbildung unterscheidet sich in einer Base. (Quelle: A Single Nucleotide Polymorphism is a change of a nucleotide at a single base-pair location on DNA. Created using OpenSCAD v2014.03 and Inkscape v0.48.5 von David Eccles via wikimedia https://commons.wikimedia.org/wiki/File:Dna-SNP.svg. CC (https://creativecommons.org/licenses/by/4.0/deed.en). Keine Veränderungen vorgenommen. sich durch den Austausch genau einer
Nukleobase. So könnte es beispielsweise sein, dass bei einer Person an besagter Stelle in der
DNA die Base Cytosin vorkommt, während bei einer anderen Person die Base Thymin liegt (siehe Abbildung 1). Ein
SNP entsteht ursprünglich durch eine Spontanmutation bei einem Individuum, wird dann aber an Nachkommen weitervererbt, und kann sich nach und nach verbreiten, sofern die genetische Veränderung keinen Nachteil zur Folge hat. Da sich neue genetische Ausprägungen allerdings nur langsam etablieren, können unterschiedliche Menschen auch unterschiedliche Ausprägungen an einem Genort haben. Eine mögliche Ausprägung an einem polymorphen Genort bezeichnet man als
Allel. Die allermeisten SNPs beinhalten den Austausch einer Base gegen eine andere Base (z. B. Cytosin gegen Thymin) und weisen daher zwei Allele auf. Die Auftretenshäufigkeit eines Allels entscheidet, ob man von einem geläufigen
SNP oder von einer seltenen Variante spricht. Bei geläufigen SNPs stellt das seltenere
Allel mindestens 1 % der Allele in der Bevölkerung.
Abbildung 1: Darstellung eines Einzelnukleotipolymorphismus (SNP). Zu sehen ist die doppelsträngige DNA in der bekannten Doppelhelixform. Der DNA Abschnitt in der oberen und unteren Abbildung unterscheidet sich in einer Base. (Quelle: A Single Nucleotide Polymorphism is a change of a nucleotide at a single base-pair location on DNA. Created using OpenSCAD v2014.03 and Inkscape v0.48.5 von David Eccles via wikimedia https://commons.wikimedia.org/wiki/File:Dna-SNP.svg. CC (https://creativecommons.org/licenses/by/4.0/deed.en). Keine Veränderungen vorgenommen. sich durch den Austausch genau einer
Nukleobase. So könnte es beispielsweise sein, dass bei einer Person an besagter Stelle in der
DNA die Base Cytosin vorkommt, während bei einer anderen Person die Base Thymin liegt (siehe Abbildung 1). Ein
SNP entsteht ursprünglich durch eine Spontanmutation bei einem Individuum, wird dann aber an Nachkommen weitervererbt, und kann sich nach und nach verbreiten, sofern die genetische Veränderung keinen Nachteil zur Folge hat. Da sich neue genetische Ausprägungen allerdings nur langsam etablieren, können unterschiedliche Menschen auch unterschiedliche Ausprägungen an einem Genort haben. Eine mögliche Ausprägung an einem polymorphen Genort bezeichnet man als
Allel. Die allermeisten SNPs beinhalten den Austausch einer Base gegen eine andere Base (z. B. Cytosin gegen Thymin) und weisen daher zwei Allele auf. Die Auftretenshäufigkeit eines Allels entscheidet, ob man von einem geläufigen
SNP oder von einer seltenen Variante spricht. Bei geläufigen SNPs stellt das seltenere
Allel mindestens 1 % der Allele in der Bevölkerung.
Molekulare Genetik in der Psychologie
Wie können SNPs Einfluss auf psychologische Prozesse und Konstrukte wie
Intelligenz nehmen? Wie bereits erwähnt stellt ein Gen den Bauplan für ein Eiweißmolekül. Wird eine Base im genetischen Code verändert, kann es daher dazu führen, dass das resultierende Eiweißmolekül anders zusammen gebaut wird oder dass sich die Anzahl der hergestellten Moleküle verändert. In der überwiegenden Mehrzahl der Fälle kann ein verändertes Eiweißmolekül seine Aufgabe im Körper immer noch wahrnehmen. Nur kann es sein, dass es durch den veränderten Bauplan besser oder schlechter funktioniert. Da es wesentlich mehr SNPs als Gene gibt, liegen auf jedem Gen eine Vielzahl polymorpher Stellen vor, deren Effekte zwar meist alle sehr klein sind, sich allerdings aufaddieren und somit zu einer Vielzahl möglicher Eiweißmoleküle führen können. Veränderungen in den Bauplänen von Eiweißmolekülen, die Aufgaben im Gehirn wie beispielsweise die Regulation von Botenstoffen oder das Wachstum von Nervenfasern haben, können dazu führen, dass Schaltkreise im Gehirn und damit auch die nervöse
Informationsverarbeitung verändert werden.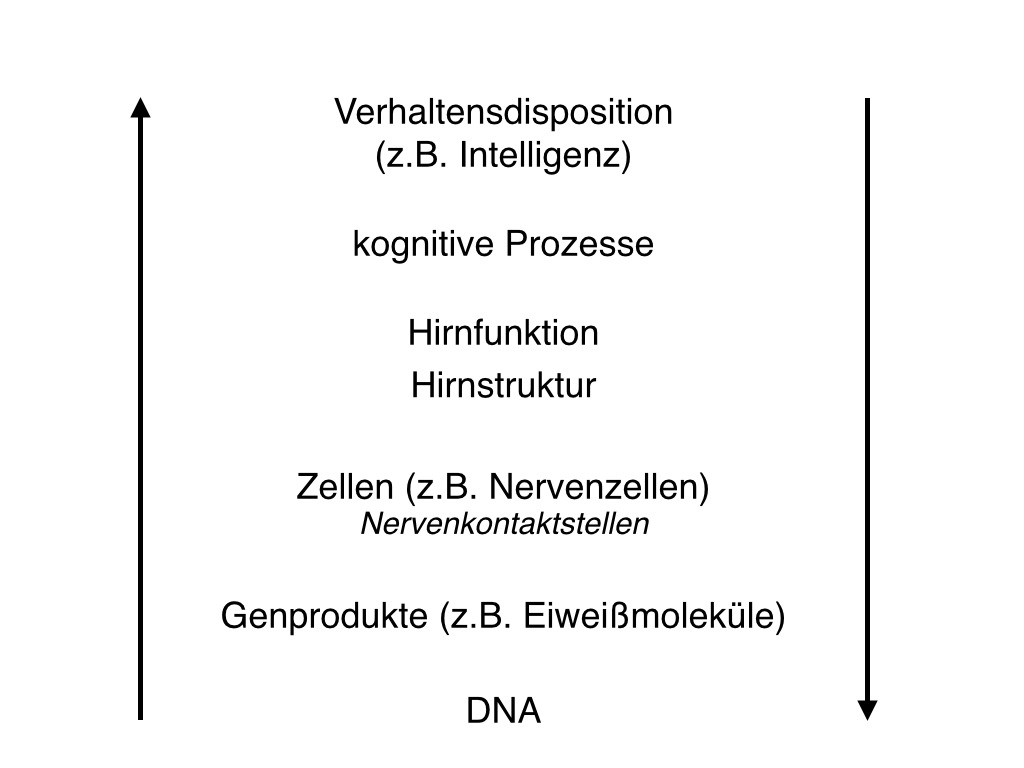 Abbildung 2: Verschiedene Betrachtungsebenen auf dem biologischen Pfad von der DNA bis zur Intelligenz. Die dargestellten Schritte sind grob aufgelöst, das heißt, dass wiederum andere Prozesse zwischen ihnen vermitteln, die nicht aufgelistet sind. © Sebastian Markett
Abbildung 2: Verschiedene Betrachtungsebenen auf dem biologischen Pfad von der DNA bis zur Intelligenz. Die dargestellten Schritte sind grob aufgelöst, das heißt, dass wiederum andere Prozesse zwischen ihnen vermitteln, die nicht aufgelistet sind. © Sebastian Markett
Viele Gene mit kleinen Effekten
Das letztendliche Ziel der molekulargenetischen Intelligenzforschung sollte es sein, genau diese neurobiologischen Pfade aufzuklären, die über verschiedene Betrachtungsebenen zwischen genetischen Unterschieden auf der einen und kognitiven Leistungsunterschieden auf der anderen Seite vermittelt (siehe Abbildung 2). Dazu ist es aber zunächst notwendig, genetische Varianten zu identifizieren, die in einem verlässlichen Zusammenhang mit der Intelligenz stehen. Doch nach wie vielen SNPs suchen wir überhaupt? Diese Frage ist nicht einfach zu beantworten, allerdings lässt sich aus einer genaueren Betrachtung der Verteilung des Merkmals „Intelligenz“ in der Bevölkerung der Schluss ziehen, dass eine Vielzahl von SNPs beteiligt sein müssen. Abbildung 3 zeigt die typische glockenförmige Verteilung, die man erhält, wenn man die Häufigkeit von verschiedenen Intelligenzausprägungen untersucht: Sehr viele Individuen haben eine mittlere Ausprägung, einige Individuen eine hohe oder niedrige und nur sehr wenige eine extreme Ausprägung. Da die Hälfte der Unterschiede im Merkmal Intelligenz auf genetische Faktoren zurückzuführen ist, würde bei der Beteiligung nur einer Genvariante keine glockenförmige Verteilung mit einem Gipfel in der Mitte, sondern eine Verteilung mit zwei Gipfeln zu erwarten sein: Der weiter links gelegene Gipfel würde Menschen vereinen, die ein Allel tragen, das zu einer geringeren Merkmalsausprägung führt, während der weiter rechts gelegene Gipfel Menschen vereinen würde, die ein Allel tragen, das die Intelligenz fördert (siehe Montag & Reuter, 2014). Je mehr SNPs zu Intelligenzunterschieden beitragen, desto geringer fällt natürlich der Beitrag eines einzelnen SNPs aus. Schätzungen gehen davon aus, dass der Beitrag eines einzelnen SNPs zu den Gesamtunterschieden zwischen Menschen unter 1 % liegt (Plomin, Kennedy & Craig, 2006). Dies bedeutet, dass es kein einzelnes „Intelligenzgen“ gibt, sondern genetisch bedingte Unterschiede im Merkmal Intelligenz von einer Vielzahl von Genen verantwortet werden.
Aus der Forschung zu anderen g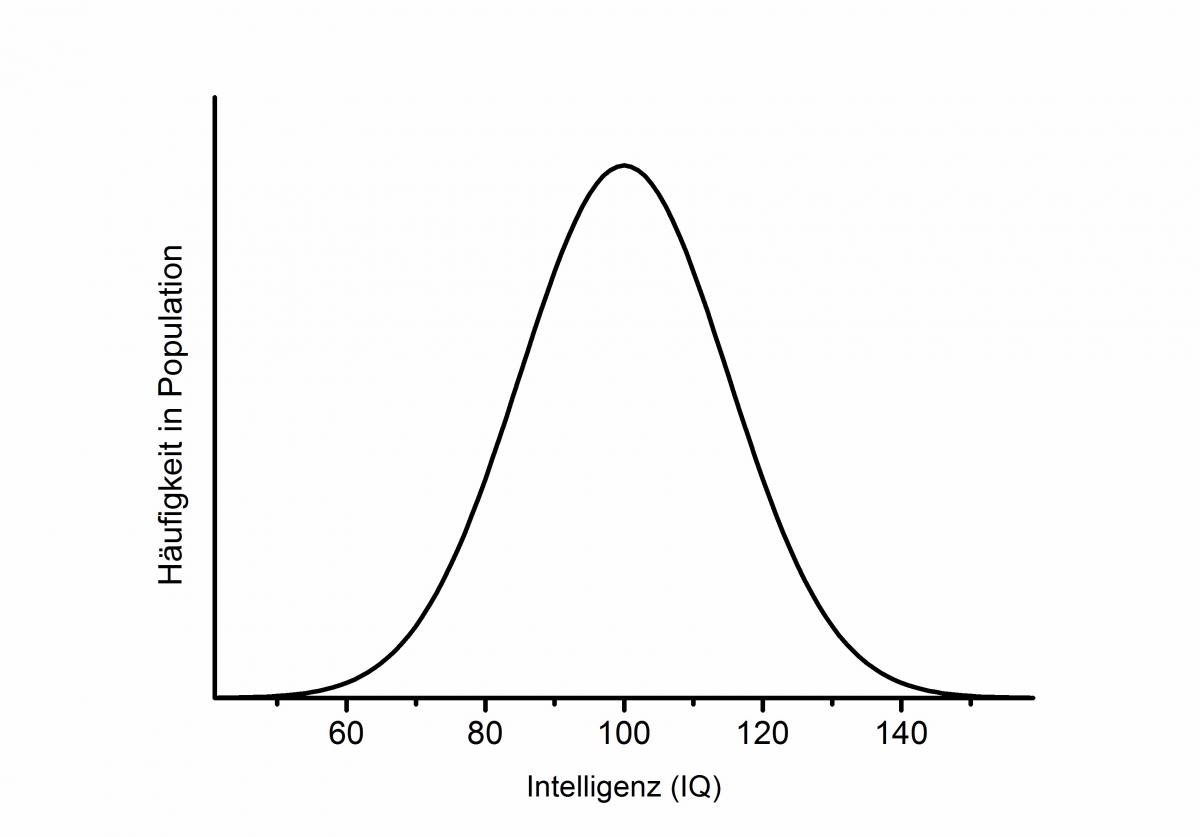 Abbildung 3: Glockenförmige Verteilung des Merkmals “Intelligenz” (ausgedrückt als Intelligenzquotient) in der Bevölkerung. © Sebastian Markettlockenförmig verteilten Merkmalen wie beispielsweise Körpergröße ist bekannt, dass bei Vorliegen einer Genvariante, die in einem begründbaren Zusammenhang zum interessierenden Merkmal steht, die Ausprägung des Merkmals leicht erhöht ist (Plomin et al., 2006). Daher ist es nicht notwendig, spezielle Versuchspersonen zu selektieren, um die Genome besonders hoch- und niedrigintelligenter Individuen zu vergleichen. Es reicht aus, in einer hinreichend großen Personenzahl die Intelligenzausprägung von TrägerInnen verschiedener Allele eines SNPs zu vergleichen. Eine Vielzahl von Studien hat in den letzten Jahren versucht, mit einem solchen Studiendesign den Zusammenhang zwischen
Intelligenz und SNPs auf verschiedenen Genen herzustellen, die aufgrund ihrer neurobiologischen Relevanz ausgewählt wurden. Diese Studien lieferten zwar mitunter Hinweise auf einzelne SNPs, allerdings konnte keines der Ergebnisse in weiteren Studien bestätigt werden (Deary, Johnson & Houlihan, 2009). Mögliche Gründe für diese negativen Befunde könnten darin liegen, dass zu wenige Versuchspersonen in den Studien untersucht wurden, um verlässliche Befunde zu erzielen, oder dass die falschen Kandidatengene ausgewählt wurden.
Abbildung 3: Glockenförmige Verteilung des Merkmals “Intelligenz” (ausgedrückt als Intelligenzquotient) in der Bevölkerung. © Sebastian Markettlockenförmig verteilten Merkmalen wie beispielsweise Körpergröße ist bekannt, dass bei Vorliegen einer Genvariante, die in einem begründbaren Zusammenhang zum interessierenden Merkmal steht, die Ausprägung des Merkmals leicht erhöht ist (Plomin et al., 2006). Daher ist es nicht notwendig, spezielle Versuchspersonen zu selektieren, um die Genome besonders hoch- und niedrigintelligenter Individuen zu vergleichen. Es reicht aus, in einer hinreichend großen Personenzahl die Intelligenzausprägung von TrägerInnen verschiedener Allele eines SNPs zu vergleichen. Eine Vielzahl von Studien hat in den letzten Jahren versucht, mit einem solchen Studiendesign den Zusammenhang zwischen
Intelligenz und SNPs auf verschiedenen Genen herzustellen, die aufgrund ihrer neurobiologischen Relevanz ausgewählt wurden. Diese Studien lieferten zwar mitunter Hinweise auf einzelne SNPs, allerdings konnte keines der Ergebnisse in weiteren Studien bestätigt werden (Deary, Johnson & Houlihan, 2009). Mögliche Gründe für diese negativen Befunde könnten darin liegen, dass zu wenige Versuchspersonen in den Studien untersucht wurden, um verlässliche Befunde zu erzielen, oder dass die falschen Kandidatengene ausgewählt wurden.
Genomweite Suche
Der technologische Fortschritt in den Lebenswissenschaften hat es in den letzten Jahren möglich und erschwinglich gemacht, das gesamte Genom nach Varianten zu durchsuchen, die statistisch zu einer Veränderung im Erscheinungsbild der Intelligenz führen. Davies et al. (2011) nutzen in einer groß angelegten Studie mit insgesamt 3 511 VersuchsteilnehmerInnen diese Technik und analysierten den Einfluss von beinahe 600 000 geläufigen SNPs auf Intelligenz. Doch auch in dieser Studie konnte für keine der untersuchten Varianten einzeln ein verlässlicher Zusammenhang gefunden werden. Das Problem einer solchen genomweiten Assoziationsstudie ist allerdings, dass sehr viele statistische Zusammenhänge zwischen einzelnen SNPs und dem interessierenden Merkmal betrachtet werden. Durch die riesige Zahl an untersuchten Zusammenhängen kann es durch Zufall zu vermeintlichen Befunden kommen, die sich in weiteren Untersuchungen nicht bestätigen lassen. Dies lässt sich an einer einfachen Analogie nachvollziehen: Wenn 600 000 Fußballspieler zum Elfmeterschießen antreten, können vereinzelt auch schlechte Elfmeterschützen vom Punkt erfolgreich sein und dann fälschlicherweise als sichere Schützen klassifiziert werden. Aus diesem Grund müssen die Ergebnisse zufallskorrigiert werden. Dies kann allerdings dazu führen, dass tatsächliche Zusammenhänge übersehen werden, da lediglich die stärksten Zusammenhänge zum interessierenden Merkmal aufgespürt werden können. Um in unserer Analogie zu bleiben: Es könnte festgelegt werden, dass nur Fußballer, die mindestens 50 Elfmeter verwandeln, als gute Elfmeterschützen klassifiziert werden, um zu verhindern, dass schlechte Schützen fälschlich ausgewählt werden. Dies bringt aber die Gefahr mit sich, dass auch manch guter Schütze nicht ausgewählt wird, weil 50 verwandelte Elfmeter eine echte Herausforderung sind. Um dieses Problem zu umgehen, bedienten sich die AutorInnen eines statistischen Tricks: Sie berechneten ein Modell, dass es ermöglicht, die Intelligenzausprägung der ProbandInnen aus allen SNPs simultan vorherzusagen. Diese Methode erlaubt zwar keinen Rückschluss mehr auf einzelne Varianten oder einzelne Gene, kann allerdings Auskunft darüber geben, ob irgendwelche der untersuchten SNPs mit dem Merkmal assoziiert sind. Diese Analyse ergab, dass 40 % der beobachteten Intelligenzunterschiede in der Stichprobe auf Basis der untersuchten SNPs vorhergesagt werden konnten. Diese Vorhersage erklärt zwar nicht den Gesamtbeitrag genetischer Faktoren von ca. 50 % (Plomin, Owen & McGuffin, 1994). Doch immerhin konnte bestätigt werden, dass häufig vorkommenden genetischen Varianten mit dem Großteil des genetischen Anteils am Merkmal Intelligenz assoziiert sind, auch wenn ihre genaue Identität im Dunkeln bleibt. Für den nicht erklärten genetischen Beitrag zur Intelligenz bestehen mindestens zwei Möglichkeiten (Gibson, 2012): So könnte es sein, dass seltene genetische Varianten, die in genomweiten Assoziationsstudien nicht berücksichtigt werden, ebenfalls ihren Beitrag leisten. Eine andere Möglichkeit wäre die Abhängigkeit einzelner genetischer Effekte von Umwelteinflüssen oder von Wechselwirkungen mit anderen Genvarianten. Dies würde dazu führen, dass einzelne SNPs nur dann einen Beitrag zur Intelligenz liefern, wenn bestimmte Rahmenbedingungen erfüllt sind. Da diese Rahmenbedingungen für verschiedene Menschen anders sein können, ist es schwierig, solche Effekte mit dem verwendeten Studiendesign aufzuspüren.
Eine aktuelle Studie (Benyamin et al., 2014) konnte den Befund von Davies und KollegInnen in einer Stichprobe von fast 18 000 Kindern bestätigten. Doch auch hier konnte nicht der gesamte genetische Anteil durch geläufige SNPs erklärt werden. Allerdings konnten die AutorInnen in dieser großen Stichprobe Genvarianten auf dem FNBP1L-Gen identifizieren, die in einem verlässlichen Zusammenhang zu allgemeinen kognitiven Funktionen zu stehen scheinen. Hinweise auf eine mögliche Rolle dieses Gens für Intelligenz hatte es schon in vorangegangen Studien gegeben, allerdings stand eine unabhängige Bestätigung noch aus. Das FNBP1L-Gen stellt den Bauplan für Eiweißmoleküle, die eine Rolle bei der Formgebung von Nervenzellen spielen. Ein genaues Erklärungsmodel für den Zusammenhang mit Intelligenz ist allerdings noch unbekannt.
Die Suche geht weiter
Wo stehen wir bei der Suche nach den molekularen Grundlagen der Intelligenz und wie soll die Suche weitergehen? Die Tatsache, dass bis jetzt keine – vielleicht mit Ausnahme von FNBP1L – verlässliche Assoziation zwischen einzelnen Genvarianten und Maßen allgemeiner kognitiver Fähigkeiten gefunden wurde, ist ernüchternd und zeigt, dass wir noch relativ am Anfang der Suche stehen. Allerdings konnten insbesondere die aktuellen genomweiten Assoziationsstudien zeigen, dass die Antwort auf die Frage nach den molekularen Grundlagen zum Großteil in geläufigen Varianten zu finden ist. Allerdings sind sehr viele Varianten mit jeweils sehr kleiner Vorhersagekraft beteiligt. Es besteht Grund für verhaltenen Optimismus, dass auf Grundlage dieser Pionierarbeiten mit größeren Stichproben und sensitiveren Statistiken genau jene Genvarianten aufgespürt werden. Des Weiteren stellen SNPs die vielleicht häufigste, aber nicht einzige Art genetischer Unterschiede dar. So werden in neuester Zeit auch beispielsweise strukturelle Unterschiede in der DNA, die mehr als eine Nukleobase betreffen, betrachtet. Diese sogenannten Kopierzahlvariationen können aus der Wiederholung oder Aussparung längerer Sequenzen aus Nukleobasen bestehen und tragen neuesten Schätzungen zufolge wesentlich zur genetischen Vielfalt bei.
Neue Strategien: Hand in Hand zur Lösung des Rätsels
Neben der Identifikation von kausal verantwortlichen genetischen Varianten ist allerdings die Aufklärung der neurobiologischen Pfade, die den statistischen Zusammenhang zwischen SNP und Intelligenz erklären, genauso wichtig. Schon jetzt besteht die Möglichkeit, nach Assoziationen zwischen Genvarianten und Merkmalen auf den in Abbildung 2 beschriebenen Betrachtungsebenen, die in einem begründbaren Zusammenhang mit Intelligenz stehen, zu suchen. Dieser Ansatz ist in der psychiatrischen Genetik weit verbreitet und wird als „Endophänotypenansatz“ bezeichnet (Gottesman & Gould, 2003). Da sehr viele vermittelnde Schritte zwischen genetischen Unterschieden und einer Verhaltensdisposition wie Intelligenz liegen, sind die Chancen, verlässliche Genassoziationen mit relevanten biologischen Merkmalen wie Hirnaktivierungsmustern oder der funktionellen und strukturellen Organisation neuronaler Schaltkreise zu finden, wesentlich höher. Außerdem bietet dieser Ansatz die Möglichkeit, die molekularen Mechanismen der Intelligenz zu erklären und über eine Beschreibung der Zusammenhänge zwischen genetischen Unterschieden und kognitiven Merkmalen hinauszugehen. Beispiele für vielversprechende Endophänotypen sind das Arbeitsgedächtnis und Organisationsprinzipien des Konnektoms (Gesamtheit neuronaler Verknüpfungen im Gehirn). Das Arbeitsgedächtnis ist ein Gedächtnissystem, das der kurzfristigen Aufrechterhaltung und Manipulation von Information dient und eine wichtige Rolle bei der Handlungsplanung und beim problemlösenden Denken spielt (Baddeley, 1986). Die neuronalen Grundlagen des Arbeitsgedächtnis sind vergleichsweise gut untersucht und es sind vielversprechende genetische Assoziationen dokumentiert (Markett, Montag & Reuter., 2010; Reuter et al., 2007; Stelzel, Basten, Montag, Reuter & Fiebach, 2009;). Das Konnektom wiederum beschreibt die Gesamtheit neuronaler Verknüpfungen im Gehirn. Es gibt Hinweise, dass Intelligenz in einem stark positiven Zusammenhang zur Informationsverarbeitungseffizienz des Konntekoms steht (van den Heuwel, Stam, Kahn & Pol, 2009). Die Gehirne hochintelligenter Menschen können verschiedene Informationen scheinbar schneller verknüpfen und verarbeiten. Molekulargenetische Studien zum Konnektom scheinen vielversprechend, stehen aber noch aus. Der Endophänotypenansatz ist eine komplementäre Strategie zu genomweite Assoziationsstudien. Gemeinsam werden sie in den kommenden Jahren die molekularen Grundlagen von Intelligenzunterschieden entschlüsseln und damit helfen, eines der zentralen Rätsel der Psychologie zu lösen.
Literatur
Baddeley, A. D. (1986). Working memory. Oxford, UK: University Press.
Benyamin, B., Pourcain, B., Davis, O. S., Davies, G., Hansell, N. K., Brion, M.-J. A., et al. (2014). Childhood intelligence is heritable, highly polygenic and associated with FNBP1L. Molecular Psychiatry, 19, 253-258.
Davies, G., Tenesa, A., Payton, A., Yang, J., Harris, S. E., Liewald, D., et al. (2011). Genome-wide association studies establish that human intelligence is highly heritable and polygenic. Molecular Psychiatry, 16, 996-1005.
Deary, I. J., Johnson, W. & Houlihan, L. M. (2009). Genetic foundations of human intelligence. Human Genetics, 126, 215-232.Montag, C. & Reuter, M. (2014). Disentangling the molecular genetic basis of personality: From monoamines to neuropeptides. Neuroscience & Biobehavioral Reviews, 43, 228-239.
Gibson, G. (2012). Rare and common variants: twenty arguments. Nature Reviews Genetics, 13, 135-145.
Gottesman, I. I. & Gould, T. D. (2003). The endophenotype concept in psychiatry: etymology and strategic intentions. The American Journal of Psychiatry, 160, 636-645.
Markett, S., Montag, C. & Reuter, M. (2010). The association between dopamine DRD2 polymorphisms and working memory capacity is modulated by a functional polymorphism on the nicotinic receptor gene CHRNA4. Journal of Cognitive Neuroscience, 22, 1944-1954.
Plomin, R., Kennedy, J. K. & Craig, I. W. (2006). The quest for quantitative trait loci associated with intelligence. Intelligence, 34, 513-526.
Plomin, R., Owen, M. J. & McGuffin, P. (1994). The genetic basis of complex human behaviors. Science, 264, 1733-1739.
Reuter, M., Esslinger, C., Montag, C., Lis, S., Gallhofer, B. & Kirsch, P. (2008). A functional variant of the tryptophan hydroxylase 2 gene impacts working memory: A genetic imaging study. Biological Psychology, 79, 111-117.
Stelzel, C., Basten, U., Montag, C., Reuter, M. & Fiebach, C. J. (2009). Effects of dopamine-related gene–gene interactions on working memory component processes. European Journal of Neuroscience, 29, 1056-1063.
van den Heuvel, M. P., Stam, C. J., Kahn, R. S. & Pol, H. E. H. (2009). Efficiency of functional brain networks and intellectual performance. The Journal of Neuroscience, 29, 7619-7624.



